
Imprintingerkrankungen
Genomisches Imprinting ist ein regulatorischer Prozess, der zu einer monoallelischen, Eltern-spezifischen Expression bestimmter Gene führt. Bislang sind ca. 100 Gene beim Menschen beschrieben, die dem genomischen Imprinting unterliegen; davon sind einige krankheitsassoziiert. Die bekanntesten liegen auf den Chromosomen 6, 7, 11, 14, 15 und 20.
Die Regulation der Eltern-spezifischen Expression ist i.d.R. an DNA-Methylierung gekoppelt, unterscheidet sich z.T. bei den verschiedenen Genen/Genclustern oder ist nur gewebsspezifisch.
Störungen dieser Regulation können genetischer oder epigenetischer Natur sein, die in cis oder in trans vorliegen, in Mosaikkonstellationen oder auch nur gewebsspezifisch vorkommen, und führen je nach betroffener Region/betroffenem Gen zu verschiedenen Imprinting-Erkrankungen.
Die Imprinting-Erkrankungen gehören alle zur Gruppe der seltenen Erkrankungen und umfassen ein breites Spektrum an klinischen Merkmalen sowie molekulargenetischen Veränderungen/Ursachen (Weitere Informationen zur Diagnostik finden Sie hier).
Allgemein sind wir sehr daran interessiert, das Wissen über die Expressions-Regulation der verschiedenen Imprintingcluster zu erweitern und zu vertiefen und diese Erkenntnisse auch in die Diagnostik zu übertragen.
Unsere Schwerpunkte liegen dabei auf
Chromosom 15q11q13
Angelman-, Prader-Willi- und Schaaf-Yang-Syndrom
Das Angelman- und das Prader-Willi-Syndrom sind neurogenetische Erkrankungen, die durch genetische und epigenetische Veränderungen auf Chromosom 15q11q13 verursacht werden. Die häufigste Ursache sind große Deletionen, die durch hochsequenzhomologe Bruchpunktregionen und die dadurch erfolgende nicht-allelische homologe Rekombination bedingt sind. Eine weitere Ursache ist die uniparentale Disomie für das Chromosom 15. Zudem gibt es eine Reihe von Patienten, bei denen eine epigenetische Veränderung ohne erkennbare zu Grunde liegende Sequenzänderung vorliegt, die Imprintingdefekte (IDs) genannt werden.
Einer unserer Forschungsschwerpunkte ist die Frage, ob es eventuell modifizierende Faktoren gibt, die zu einem ID prädisponieren können.
Eine weitere Erkrankung, die durch ein Gen in der Region 15q11q13 verursacht wird, ist das Schaaf-Yang-Syndrom. Patienten mit diesem Syndrom zeigen einen mit dem Prader-Willi-Syndrom stark überlappenden klinischen Phänotyp. Allerdings führen nur trunkierende Varianten auf dem aktiven paternalen Allel des MAGEL2-Gens zu einer Erkrankung. Wir und andere konnten zeigen, dass Deletionen des gesamten Gens, die nicht das Imprinting-Center der Region betreffen, nicht zu einem solchen Phänotyp führen.
Chromosom 14q32
Temple- und Kagami-Ogata-Syndrom
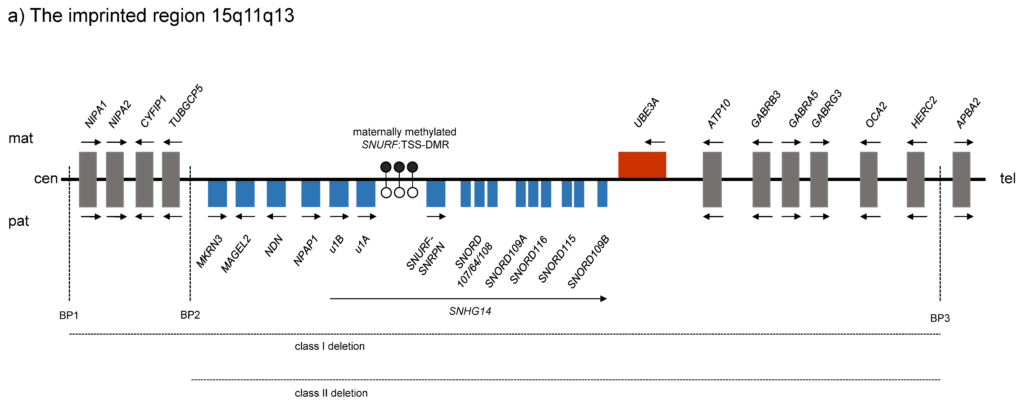
Die chromosomale Region 14q32 beinhaltet ein Imprintingcluster, das drei DMRs und zahlreiche maternal oder paternal exprimierte Gene umfasst. Dabei fungieren zwei der drei DMRs als Imprinting-Center (IC), die für die Regulation der elternspezifischen Expression unerlässlich sind. Wir und andere konnten zeigen, dass diese beiden ICs dabei in einer hierarchischen Struktur agieren. Zur Funktion der dritten DMR, die reziprok zu den beiden anderen methyliert ist, ist bisher noch nicht so viel bekannt und daher Teil unserer Forschung.
Als Ursache für ein Temple- oder Kagami-Ogata-Syndrom wurden von uns und anderen bereits verschiedene Deletionen beschrieben. Die detaillierte Charakterisierung solcher Deletionen ist dabei zur weiteren Erforschung der Regulationsmechanismen innerhalb des Clusters auf 14q32 als auch für die Diagnostik wichtig.
Chromosom 11p15.5
Beckwith-Wiedemann- und Silver-Russell-Syndrom
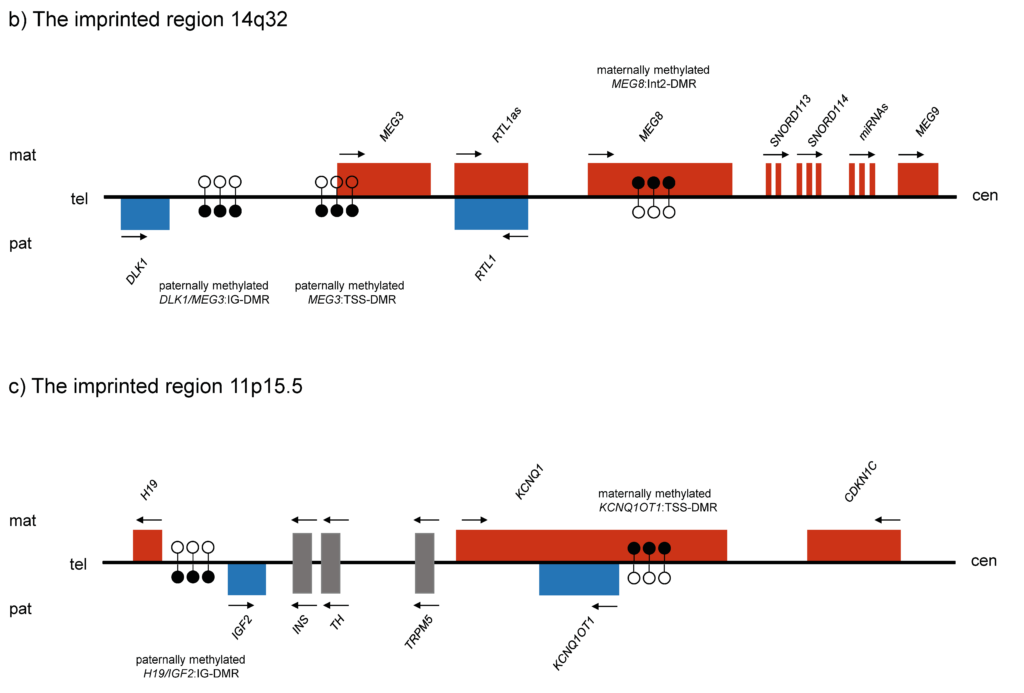
Das Beckwith-Wiedemann- (BWS) und das Silver-Russell-Syndrom (SRS) werden in der überwiegenden Mehrheit der Fälle durch Imprintingdefekte im Mosaik verursacht. Bei einem Teil der Patienten liegt jedoch eine Sequenzveränderung zu Grunde, die die Methylierungsveränderung bedingt. Hierzu gehören zum Beispiel Mikrodeletionen und Einzelnukleotidaustausche innerhalb der Imprintingkontrollregion 1 (ICR1), die die CTCF- bzw. SOX2-Bindestellen betreffen. Unterschiedliche Deletionen führen hierbei zu unterschiedlich starken Methylierungsveränderungen.
Zu berücksichtigen sind auch Deletionen das Gen KCNQ1 betreffend, die ebenfalls zu einer Methylierungsveränderung führen aber nur bei der ICR2.
Ein weiterer Aspekt, der gerade bei Patienten mit diesen Syndromen eine Rolle spielt, sind sogenannte Multilocus-Imprintingdefekte, bei denen mehrere dem Imprinting unterliegende Loci eine Methylierungsveränderung aufweisen. In manchen Fällen konnten hier pathogene Varianten bei den Müttern der Patienten in einer Reihe von maternalen Effekt-Genen nachgewiesen werden. Diese Gene sind am sogenannten subkortikalen maternalen Komplex (SCMC) beteiligt. Dabei ist der genaue Mechanismus, wie dies zu den Methylierungsveränderungen an multiplen Loci führt, noch nicht geklärt. Auch sind trotz mehrerer Krankheitsassoziierter Loci, die eine Methylierungsveränderung aufweisen, in der Regel der BWS bzw. SRS Phänotyp vorherrschend. Wieso dies der Fall ist, ist ebenfalls Gegenstand weiterer Forschung.
Ansprechpartner/Kontakt
Dr. rer. nat.
Jasmin Beygo
Dr. rer. nat.
Deniz Kanber
Dr. rer. nat.
Sandra Ueberberg
Ausgewählte Publikationen
Beygo J, Kanber D, Eggermann T, Begemann M
Molecular testing for imprinting disorders.
Medizinische Genetik 2020, 32:305-319.
Beygo J, Russo S, Tannorella P, Santen GWE, Dufke A, Schlaich E, Eggermann T
Prenatal testing for imprinting disorders: A laboratory perspective.
Prenat Diagn 2023, 43:973-982.
Marbach F, Elgizouli M, Rech M, Beygo J, Erger F, Velmans C, Stumpel C, Stegmann APA, Beck-Wodl S, Gillessen-Kaesbach G, Horsthemke B, Schaaf CP, Kuechler A
The adult phenotype of Schaaf-Yang syndrome.
Orphanet J Rare Dis 2020, 15:294.
Beygo J, Grosser C, Kaya S, Mertel C, Buiting K, Horsthemke B
Common genetic variation in the Angelman syndrome imprinting centre affects the imprinting of chromosome 15.
Eur J Hum Genet 2020, 28:835-839.
Beygo J*, Buiting K, Ramsden SC, Ellis R, Clayton-Smith J, Kanber D*
Update of the EMQN/ACGS best practice guidelines for molecular analysis of Prader-Willi and Angelman syndromes.
Eur J Hum Genet 2019, 27:1326-1340.
(*equal contribution)
Le Fevre A, Beygo J, Silveira C, Kamien B, Clayton-Smith J, Colley A, Buiting K, Dudding-Byth T
Atypical Angelman syndrome due to a mosaic imprinting defect: Case reports and review of the literature.
Am J Med Genet A 2017, 173:753-757.
Buiting K, Di Donato N, Beygo J, Bens S, von der Hagen M, Hackmann K, Horsthemke B.
Clinical phenotypes of MAGEL2 mutations and deletions.
Orphanet J Rare Dis 2014; 9:40.
Gillessen-Kaesbach G, Albrecht B, Eggermann T, Elbracht M, Mitter D, Morlot S, van Ravenswaaij-Arts CMA, Schulz S, Strobl-Wildemann G, Buiting K, Beygo J
Molecular and clinical studies in 8 patients with Temple syndrome.
Clin Genet 2018, 93:1179-1188.
Beygo J, Kuchler A, Gillessen-Kaesbach G, Albrecht B, Eckle J, Eggermann T, Gellhaus A, Kanber D, Kordass U, Ludecke HJ, Purmann S, Rossier E, van de Nes J, van der Werf IM, Wenzel M, Wieczorek D, Horsthemke B, Buiting K
New insights into the imprinted MEG8-DMR in 14q32 and clinical and molecular description of novel patients with Temple syndrome.
Eur J Hum Genet 2017.
Bens S, Kolarova J, Gillessen-Kaesbach G, Buiting K, Beygo J, Caliebe A, Ammerpohl O, Siebert R.
The differentially methylated region of MEG8 is hypermethylated in patients with Temple syndrome.
Epigenomics 2015.
Beygo J, Elbracht M, de Groot K, Begemann M, Kanber D, Platzer K, Gillessen-Kaesbach G, Vierzig A, Green A, Heller R, Buiting K, Eggermann T.
Novel deletions affecting the MEG3-DMR provide further evidence for a hierarchical regulation of imprinting in 14q32.
Eur J Hum Genet 2015, 23:180-188.
Schwaibold EMC, Beygo J, Obeid K, Jauch A, Hinderhofer K, Moog U
A boy with Silver-Russell syndrome and Sotos syndrome.
Am J Med Genet A 2021, 185:549-554.
Coktu S, Spix C, Kaiser M, Beygo J, Kleinle S, Bachmann N, Kohlschmidt N, Prawitt D, Beckmann A, Klaes R, Nevinny-Stickel-Hinzpeter C, Dohnert S, Kraus C, Kadgien G, Vater I, Biskup S, Kutsche M, Kohlhase J, Eggermann T, Zenker M, Kratz CPCancer incidence and spectrum among children with genetically confirmed Beckwith-Wiedemann spectrum in Germany: a retrospective cohort study.
Br J Cancer 2020.
Beygo J, Burger J, Strom TM, Kaya S, Buiting K
Disruption of KCNQ1 prevents methylation of the ICR2 and supports the hypothesis that its transcription is necessary for imprint establishment.
Eur J Hum Genet 2019, 27:903-908.
Beygo J, Joksic I, Strom TM, Ludecke HJ, Kolarova J, Siebert R, Mikovic Z, Horsthemke B, Buiting K
A maternal deletion upstream of the imprint control region 2 in 11p15 causes loss of methylation and familial Beckwith-Wiedemann syndrome.
Eur J Hum Genet 2016, 24:1280-1286.
Beygo J, Citro V, Sparago A, De Crescenzo A, Cerrato F, Heitmann M, Rademacher K, Guala A, Enklaar T, Anichini C, Cirillo Silengo M, Graf N, Prawitt D, Cubellis MV, Horsthemke B, Buiting K, Riccio A.
The molecular function and clinical phenotype of partial deletions of the IGF2/H19 imprinting control region depends on the spatial arrangement of the remaining CTCF-binding sites.
Hum Mol Genet 2013; 22:544-57.
Berland S, Appelback M, Bruland O, Beygo J, Buiting K, Mackay DJ, Karen Temple I, Houge G.
Evidence for anticipation in Beckwith-Wiedemann syndrome.
Eur J Hum Genet 2013; 21:1344-8.
Mackay DJG, Gazdagh G, Monk D, Brioude F, Giabicani E, Krzyzewska IM, Kalish JM, Maas SM, Kagami M, Beygo J, Kahre T, Tenorio-Castano J, Ambrozaityte L, Burnyte B, Cerrato F, Davies JH, Ferrero GB, Fjodorova O, Manero-Azua A, Pereda A, Russo S, Tannorella P, Temple KI, Ounap K, Riccio A, de Nanclares GP, Maher ER, Lapunzina P, Netchine I, Eggermann T, et al:
Multi-locus imprinting disturbance (MLID): interim joint statement for clinical and molecular diagnosis.
Clin Epigenetics 2024, 16:99.
Mackay D, Bliek J, Kagami M, Tenorio-Castano J, Pereda A, Brioude F, Netchine I, Papingi D, de Franco E, Lever M, Sillibourne J, Lombardi P, Gaston V, Tauber M, Diene G, Bieth E, Fernandez L, Nevado J, Tumer Z, Riccio A, Maher ER, Beygo J, Tannorella P, Russo S, de Nanclares GP, Temple IK, Ogata T, Lapunzina P, Eggermann T
First step towards a consensus strategy for multi-locus diagnostic testing of imprinting disorders.
Clin Epigenetics 2022, 14:143.
Begemann M, Rezwan FI, Beygo J, Docherty LE, Kolarova J, Schroeder C, Buiting K, Chokkalingam K, Degenhardt F, Wakeling EL, Kleinle S, Gonzalez Fassrainer D, Oehl-Jaschkowitz B, Turner CLS, Patalan M, Gizewska M, Binder G, Bich Ngoc CT, Chi Dung V, Mehta SG, Baynam G, Hamilton-Shield JP, Aljareh S, Lokulo-Sodipe O, Horton R, Siebert R, Elbracht M, Temple IK, Eggermann T, Mackay DJG
Maternal variants in NLRP and other maternal effect proteins are associated with multilocus imprinting disturbance in offspring.
J Med Genet 2018.
Bens S, Kolarova J, Beygo J, Buiting K, Caliebe A, Eggermann T, Gillessen-Kaesbach G, Prawitt D, Thiele-Schmitz S, Begemann M, Enklaar T, Gutwein J, Haake A, Paul U, Richter J, Soellner L, Vater I, Monk D, Horsthemke B, Ammerpohl O, Siebert R
Phenotypic spectrum and extent of DNA methylation defects associated with multilocus imprinting disturbances.
Epigenomics 2016, 8:801-816.
